BiaPy, una herramienta de inteligencia artificial accesible para el análisis de imágenes biomédicas
Un equipo internacional de investigadores e investigadoras liderados por Ignacio Arganda (Universidad del País Vasco -UPV/EHU, Ikerbasque, Donostia International Physics Center, e Instituto Biofisika) y Arrate Muñoz Barrutia (Universidad Carlos III de Madrid, Instituto de Investigación Sanitaria Gregorio Marañón) ha desarrollado BiaPy, una plataforma de inteligencia artificial de código abierto que facilita el análisis de imágenes biomédicas mediante técnicas de aprendizaje profundo o deep learning. El trabajo ha sido publicado en la prestigiosa revista Nature Methods.
El análisis de imágenes es una herramienta esencial en biomedicina, utilizada para estudiar estructuras celulares, tejidos y órganos en múltiples disciplinas. Sin embargo, aplicar inteligencia artificial para analizar estas imágenes ha estado tradicionalmente reservado a expertos en programación y ciencia de datos. BiaPy rompe esa barrera al ofrecer una plataforma fácil de usar que permite aplicar modelos avanzados de IA sin necesidad de conocimientos técnicos especializados.
"El objetivo de BiaPy es democratizar el acceso a la inteligencia artificial en bioimagen, permitiendo que más científicos y profesionales de la salud puedan aprovechar su potencial sin necesidad de conocimientos avanzados en programación o aprendizaje automático", explica Daniel Franco, primer autor del estudio y actualmente investigador postdoctoral en el MRC Laboratory of Molecular Biology y Cambridge University (Reino Unido).
BiaPy permite realizar diferentes tipos de análisis sobre imágenes científicas, como identificar automáticamente células u otras estructuras biológicas, contar elementos, clasificar muestras según su apariencia, o mejorar la calidad de las imágenes para ver detalles más finos. Todo ello puede hacerse tanto con imágenes en dos dimensiones como con imágenes tridimensionales obtenidas mediante diferentes técnicas de microscopía. Además, BiaPy ha sido diseñado para ser eficiente y escalable: puede trabajar con volúmenes de datos muy variados, desde unas pocas imágenes pequeñas hasta terabytes de información, como los que se generan al escanear tejidos u órganos completos.
La herramienta se basa en el uso de “modelos de inteligencia artificial”, que son algoritmos entrenados para reconocer patrones en imágenes, de forma similar a cómo el ojo humano puede identificar formas o colores. Para crear un modelo, primero se le enseña con ejemplos: por ejemplo, imágenes donde las células ya han sido marcadas manualmente. Con suficiente entrenamiento, el modelo aprende a hacer estas tareas automáticamente, incluso en imágenes nuevas que nunca ha visto antes.
“Además, BiaPy está integrada en el BioImage Model Zoo (bioimage.io), una base de datos donde investigadores de todo el mundo comparten modelos ya entrenados. Gracias a esta integración, los usuarios de BiaPy pueden reutilizar modelos existentes para nuevas imágenes o entrenar sus propios modelos de manera sencilla,” explica Arrate Muñoz, co-autora senior del trabajo y miembro del consorcio europeo Al4Life que ha desarrollado el BioImage Model Zoo.
Esta herramienta ya se está utilizando en proyectos científicos avanzados. Un ejemplo es CartoCell, una solución software desarrollada en colaboración con el laboratorio coordinado por Luis M. Escudero (Instituto de Biomedicina de Sevilla (Hospital Universitario Virgen del Rocío/CSIC/Universidad de Sevilla). CartoCell analiza imágenes de microscopía para revelar patrones escondidos en la forma y distribución de las células dentro de tejidos epiteliales en 3D de diferentes organismos.
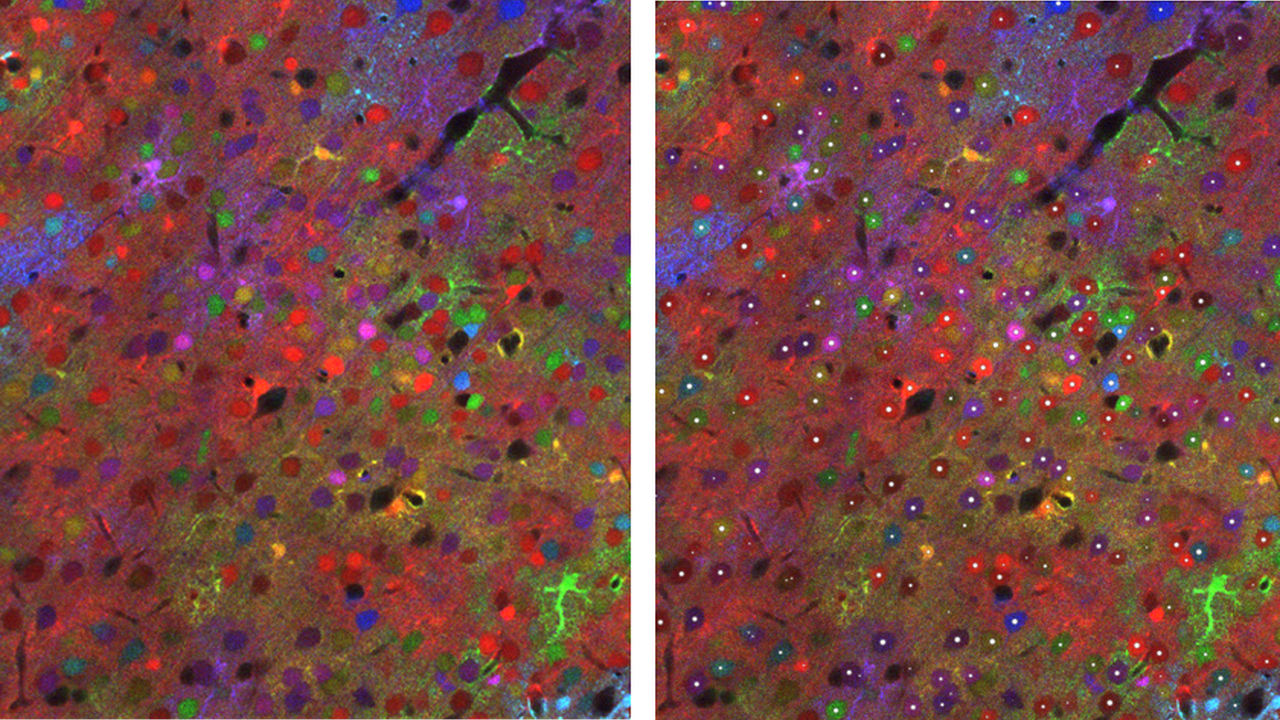
Otro caso destacado es su aplicación en colaboración con los laboratorios de Emmanuel Beaurepaire (École Polytechnique, Francia) y Jean Livet (Institut de la Vision, París). Estos grupos han desarrollado una técnica de microscopía llamada ChroMS, que permite obtener enormes imágenes tridimensionales de cerebros completos usando colores fluorescentes generados por proteínas de medusas y corales. BiaPy se utiliza para detectar automáticamente cada célula en estas imágenes a gran escala, incluso en zonas densamente pobladas del cerebro, lo que permite estudiar el desarrollo cerebral reconstruyendo el linaje de las células a partir de sus colores y posiciones tridimensionales.
Como herramienta de acceso libre, BiaPy está disponible gratuitamente para la comunidad científica, fomentando la colaboración y la mejora continua del software. Puede utilizarse en ordenadores personales o en servidores con múltiples tarjetas gráficas, así como en la nube. Su instalación es sencilla y garantiza que los experimentos puedan repetirse fácilmente en distintos entornos, promoviendo la ciencia abierta y reproducible.
En palabras de Ignacio Arganda, autor senior del trabajo, “el desarrollo de BiaPy representa un paso importante hacia la democratización de la visión artificial avanzada en microscopía. Su diseño accesible y su enfoque en la colaboración abierta reducen las barreras técnicas, facilitando que más investigadores y profesionales de la salud puedan aplicar visión artificial en sus estudios. Su compatibilidad con diversos entornos computacionales y su naturaleza de código abierto lo convierten en una plataforma con gran potencial para impulsar la innovación y acelerar el descubrimiento científico”.
BiaPy: accessible deep learning on bioimages
Daniel Franco-Barranco, Jesús A. Andrés-San Román, Ivan Hidalgo-Cenalmor, Lenka Backová, Aitor González-Marfil, Clément Caporal, Anatole Chessel, Pedro Gómez-Gálvez, Luis M. Escudero, Donglai Wei, Arrate Muñoz-Barrutia, and Ignacio Arganda-Carreras
Nature Methods 22, 4 (2025)
DOI: 10.1038/s41592-025-02699-y
